Isoforma de proteína
Uma isoforma de proteína, ou “variante de proteína”,[1] é um membro de um conjunto de proteínas altamente semelhantes que se originam de um único gene ou família de genes e são o resultado de diferenças genéticas.[2] Embora muitas desempenhem funções biológicas iguais ou semelhantes, algumas isoformas têm funções exclusivas. Um conjunto de isoformas de proteínas pode ser formado por splicings alternativos, uso variável do promotor ou outras modificações pós-transcricionais de um único gene; as modificações pós-tradução geralmente não são consideradas. Por meio de mecanismos de splicing de RNA, o mRNA tem a capacidade de selecionar diferentes segmentos codificadores de proteínas (éxons) de um gene, ou mesmo diferentes partes de éxons do RNA para formar diferentes sequências de mRNA. Cada sequência única produz uma forma específica de uma proteína.
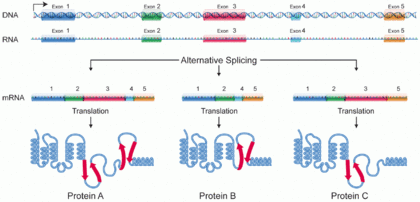
A descoberta de isoformas poderia explicar a discrepância entre o pequeno número de regiões codificadoras de proteínas dos genes revelados pelo Projeto Genoma Humano e a grande diversidade de proteínas observada em um organismo: diferentes proteínas codificadas pelo mesmo gene poderiam aumentar a diversidade do proteoma. As isoformas em nível de RNA são prontamente caracterizadas por estudos de transcrição de cDNA. Muitos genes humanos possuem isoformas de splicing alternativo confirmadas. Estima-se que cerca de 100.000 marcadores de sequências expressas (ESTs) possam ser identificados em humanos.[1] As isoformas em nível de proteína podem se manifestar na deleção de domínios inteiros ou loops mais curtos, geralmente localizados na superfície da proteína.[3]
Definição
editarUm único gene tem a capacidade de produzir várias proteínas que diferem tanto na estrutura quanto na composição;[4][5] esse processo é regulado pelo splicing alternativo do mRNA, embora não esteja claro até que ponto esse processo afeta a diversidade do proteoma humano, já que a abundância de isoformas de transcrição de mRNA não está necessariamente correlacionada com a abundância de isoformas de proteínas.[6] As comparações de estruturas tridimensionais de proteínas podem ser usadas para ajudar a determinar quais isoformas, se houver, representam produtos proteicos funcionais, e a estrutura da maioria das isoformas no proteoma humano foi prevista pelo programa AlphaFold e divulgada publicamente.[7] A especificidade das isoformas traduzidas é derivada da estrutura/função da proteína, bem como do tipo de célula e do estágio de desenvolvimento durante o qual elas são produzidas.[4][5] A determinação da especificidade se torna mais complicada quando uma proteína tem várias subunidades e cada subunidade tem várias isoformas.
Por exemplo, a proteína quinase ativada por AMP (AMPK), uma enzima que desempenha diferentes funções nas células humanas, tem 3 subunidades:[8]
- α, domínio catalítico, tem duas isoformas: α1 e α2, que são codificadas por PRKAA1 [en] e PRKAA2 [en]
- β, domínio regulatório, tem duas isoformas: β1 e β2, que são codificadas por PRKAB1 [en] e PRKAB2 [en]
- γ, domínio regulatório, tem três isoformas: γ1, γ2 e γ3, que são codificadas por PRKAG1 [en], PRKAG2 [en] e PRKAG3 [en]
No músculo esquelético humano, a forma preferida é α2β2γ1.[8] Mas no fígado humano, a forma mais abundante é α1β2γ1.[8]
Mecanismo
editarOs principais mecanismos que produzem isoformas de proteínas são o splicing alternativo e o uso de promotores variáveis, embora as modificações decorrentes de alterações genéticas, como mutações e polimorfismos, às vezes também sejam consideradas isoformas distintas.[9]
O splicing alternativo é o principal processo de modificação pós-transcricional que produz isoformas de transcrição de mRNA e é um dos principais mecanismos moleculares que podem contribuir para a diversidade de proteínas.[5] O spliceossoma, uma grande ribonucleoproteína, é a máquina molecular dentro do núcleo responsável pela clivagem e ligação do RNA, removendo segmentos não codificadores de proteínas (íntrons).[10]
Como o splicing é um processo que ocorre entre a transcrição e a tradução, seus efeitos primários têm sido estudados principalmente por meio de técnicas genômicas - por exemplo, análises de microarranjo e sequenciamento de RNA têm sido usados para identificar transcritos com splicing alternativo e medir suas abundâncias.[9] A abundância de transcritos é frequentemente usada como um indicador da abundância de isoformas de proteína, embora experimentos proteômicos usando eletroforese em gel e espectrometria de massa tenham demonstrado que a correlação entre as contagens de transcritos e proteínas é frequentemente baixa e que uma isoforma de proteína é geralmente dominante.[11] Um estudo de 2015 afirma que a causa dessa discrepância provavelmente ocorre após a tradução, embora o mecanismo seja essencialmente desconhecido.[12] Consequentemente, embora o splicing alternativo tenha sido implicado como um importante elo entre variação e doença, não há evidências conclusivas de que ele atue principalmente produzindo novas isoformas de proteína.[11]
O splicing alternativo geralmente descreve um processo rigidamente regulado no qual os transcritos alternativos são intencionalmente gerados pelo mecanismo de splicing. No entanto, esses transcritos também são produzidos por erros de splicing em um processo chamado de “splicing ruidoso” e também são potencialmente traduzidos em isoformas de proteínas. Embora se acredite que cerca de 95% dos genes multiexônicos tenham splicing alternativo, um estudo sobre splicing ruidoso observou que a maioria dos diferentes transcritos de baixa abundância são ruídos e prevê que a maioria das isoformas alternativas de transcritos e proteínas presentes em uma célula não são funcionalmente relevantes.[13]
Outras etapas regulatórias transcricionais e pós-transcricionais também podem produzir diferentes isoformas de proteínas.[14] O uso variável do promotor ocorre quando a maquinaria transcricional de uma célula (RNA polimerase, fatores de transcrição e outras enzimas) inicia a transcrição em diferentes promotores - a região do DNA próxima a um gene que serve como local de ligação inicial - resultando em transcrições e isoformas de proteínas ligeiramente modificadas.
Características
editarEm geral, uma isoforma de proteína é rotulada como a sequência canônica com base em critérios como sua prevalência e semelhança com sequências ortólogas - ou funcionalmente análogas - em outras espécies.[15] Presume-se que as isoformas tenham propriedades funcionais semelhantes, pois a maioria tem sequências semelhantes e compartilha alguns ou a maioria dos éxons com a sequência canônica. No entanto, algumas isoformas apresentam uma divergência muito maior (por exemplo, por meio de trans-splicing) e podem compartilhar poucos ou nenhum éxon com a sequência canônica. Além disso, elas podem ter efeitos biológicos diferentes - por exemplo, em um caso extremo, a função de uma isoforma pode promover a sobrevivência da célula, enquanto outra promove a morte da célula - ou podem ter funções básicas semelhantes, mas diferem em sua localização subcelular.[16] Um estudo de 2016, no entanto, caracterizou funcionalmente todas as isoformas de 1.492 genes e determinou que a maioria das isoformas se comporta como “aloformas funcionais”. Os autores chegaram à conclusão de que as isoformas se comportam como proteínas distintas depois de observar que a função da maioria das isoformas não se sobrepunha.[17] Como o estudo foi realizado em células in vitro, não se sabe se as isoformas no proteoma humano expresso compartilham essas características. Além disso, como a função de cada isoforma geralmente deve ser determinada separadamente, a maioria das isoformas identificadas e previstas ainda tem funções desconhecidas.
Conceitos relacionados
editarGlicoforma
editarUma glicoforma é uma isoforma de uma proteína que difere apenas com relação ao número ou ao tipo de glicano ligado.[18] As glicoproteínas geralmente consistem em várias glicoformas diferentes, com alterações no sacarídeo ou oligossacarídeo ligado. Essas modificações podem resultar de diferenças na biossíntese durante o processo de glicosilação ou devido à ação de beta-glicosidases ou glicosiltransferases. As glicoformas podem ser detectadas por meio de análise química detalhada de glicoformas separadas, mas são mais convenientemente detectadas por meio de reação diferencial com lectinas, como na cromatografia de afinidade [en] de lectina e na eletroforese de afinidade [en] de lectina. Exemplos típicos de glicoproteínas que consistem em glicoformas são as proteínas plasmáticas [en], como orosomucoide [en], alfa 1 antitripsina e haptoglobina. Uma variação incomum de glicoforma é observada na molécula de adesão celular neuronal (NCAM), envolvendo ácidos polissiálicos [en] (PSA).
Exemplos
editar- G-actina: apesar de sua natureza conservada, ela tem um número variável de isoformas (pelo menos seis em mamíferos).
- Creatina quinase: sua presença no sangue pode ser usada como auxílio no diagnóstico de infarto do miocárdio. Existe em três isoformas.
- Hialuronan sintase [en]: enzima responsável pela produção de hialuronan. Tem três isoformas em células de mamíferos.
- UDP-glucuronosiltransferase: superfamília de enzimas responsável pela via de desintoxicação de muitos medicamentos, poluentes ambientais e compostos endógenos tóxicos. Tem 16 isoformas conhecidas codificadas no genoma humano.[19]
- G6PDA: a proporção normal de isoformas ativas nas células de qualquer tecido é de 1:1, compartilhada com a G6PDG. Essa é exatamente a proporção normal de isoformas na hiperplasia. Apenas uma dessas isoformas é encontrada durante a neoplasia.[20]
- Monoamina oxidase: uma família de enzimas que catalisa a oxidação de monoaminas. Existe em duas isoformas, MAO-A e MAO-B.
Referências
- ↑ a b Brett D, Pospisil H, Valcárcel J, Reich J, Bork P (Janeiro de 2002). «Alternative splicing and genome complexity». Nature Genetics. 30 (1): 29–30. PMID 11743582. doi:10.1038/ng803
- ↑ Schlüter H, Apweiler R, Holzhütter HG, Jungblut PR (Setembro de 2009). «Finding one's way in proteomics: a protein species nomenclature». Chemistry Central Journal. 3. 11 páginas. PMC 2758878 . PMID 19740416. doi:10.1186/1752-153X-3-11
- ↑ Kozlowski, L.; Orlowski, J.; Bujnicki, J. M. (2012). «Structure Prediction for Alternatively Spliced Proteins». Alternative pre-mRNA Splicing. [S.l.: s.n.] 582 páginas. ISBN 9783527636778. doi:10.1002/9783527636778.ch54
- ↑ a b Andreadis A, Gallego ME, Nadal-Ginard B (1 de janeiro de 1987). «Generation of protein isoform diversity by alternative splicing: mechanistic and biological implications». Annual Review of Cell Biology. 3 (1): 207–42. PMID 2891362. doi:10.1146/annurev.cb.03.110187.001231
- ↑ a b c Breitbart RE, Andreadis A, Nadal-Ginard B (1 de janeiro de 1987). «Alternative splicing: a ubiquitous mechanism for the generation of multiple protein isoforms from single genes». Annual Review of Biochemistry. 56 (1): 467–95. PMID 3304142. doi:10.1146/annurev.bi.56.070187.002343
- ↑ Liu Y, Beyer A, Aebersold R (Abril de 2016). «On the Dependency of Cellular Protein Levels on mRNA Abundance». Cell. 165 (3): 535–50. PMID 27104977. doi:10.1016/j.cell.2016.03.014 . hdl:20.500.11850/116226
- ↑ Sommer, Markus J.; Cha, Sooyoung; Varabyou, Ales; Rincon, Natalia; Park, Sukhwan; Minkin, Ilia; Pertea, Mihaela; Steinegger, Martin; Salzberg, Steven L. (15 de dezembro de 2022). «Structure-guided isoform identification for the human transcriptome». eLife (em inglês). 11: e82556. PMC 9812405 . PMID 36519529. doi:10.7554/eLife.82556
- ↑ a b c Dasgupta B, Chhipa RR (Março de 2016). «Evolving Lessons on the Complex Role of AMPK in Normal Physiology and Cancer». Trends in Pharmacological Sciences (em inglês). 37 (3): 192–206. PMC 4764394 . PMID 26711141. doi:10.1016/j.tips.2015.11.007
- ↑ a b Kornblihtt AR, Schor IE, Alló M, Dujardin G, Petrillo E, Muñoz MJ (Março de 2013). «Alternative splicing: a pivotal step between eukaryotic transcription and translation». Nature Reviews Molecular Cell Biology (em inglês). 14 (3): 153–65. PMID 23385723. doi:10.1038/nrm3525. hdl:11336/21049
- ↑ Lee Y, Rio DC (1 de janeiro de 2015). «Mechanisms and Regulation of Alternative Pre-mRNA Splicing». Annual Review of Biochemistry. 84 (1): 291–323. PMC 4526142 . PMID 25784052. doi:10.1146/annurev-biochem-060614-034316
- ↑ a b Tress ML, Abascal F, Valencia A (Fevereiro de 2017). «Alternative Splicing May Not Be the Key to Proteome Complexity». Trends in Biochemical Sciences. 42 (2): 98–110. PMC 6526280 . PMID 27712956. doi:10.1016/j.tibs.2016.08.008
- ↑ Battle A, Khan Z, Wang SH, Mitrano A, Ford MJ, Pritchard JK, Gilad Y (Fevereiro de 2015). «Genomic variation. Impact of regulatory variation from RNA to protein». Science. 347 (6222): 664–7. PMC 4507520 . PMID 25657249. doi:10.1126/science.1260793
- ↑ Pickrell JK, Pai AA, Gilad Y, Pritchard JK (Dezembro de 2010). «Noisy splicing drives mRNA isoform diversity in human cells». PLOS Genetics. 6 (12): e1001236. PMC 3000347 . PMID 21151575. doi:10.1371/journal.pgen.1001236
- ↑ Smith LM, Kelleher NL (Março de 2013). «Proteoform: a single term describing protein complexity». Nature Methods (em inglês). 10 (3): 186–7. PMC 4114032 . PMID 23443629. doi:10.1038/nmeth.2369
- ↑ Li HD, Menon R, Omenn GS, Guan Y (Dezembro de 2014). «Revisiting the identification of canonical splice isoforms through integration of functional genomics and proteomics evidence» (PDF). Proteomics. 14 (23–24): 2709–18. PMC 4372202 . PMID 25265570. doi:10.1002/pmic.201400170
- ↑ Sundvall M, Veikkolainen V, Kurppa K, Salah Z, Tvorogov D, van Zoelen EJ, Aqeilan R, Elenius K (Dezembro de 2010). «Cell death or survival promoted by alternative isoforms of ErbB4». Molecular Biology of the Cell. 21 (23): 4275–86. PMC 2993754 . PMID 20943952. doi:10.1091/mbc.E10-04-0332
- ↑ Yang X, Coulombe-Huntington J, Kang S, Sheynkman GM, Hao T, Richardson A, et al. (Fevereiro de 2016). «Widespread Expansion of Protein Interaction Capabilities by Alternative Splicing». Cell. 164 (4): 805–17. PMC 4882190 . PMID 26871637. doi:10.1016/j.cell.2016.01.029
- ↑ «Protein Isoform - an overview | ScienceDirect Topics». www.sciencedirect.com. Consultado em 25 de outubro de 2024
- ↑ Barre L, Fournel-Gigleux S, Finel M, Netter P, Magdalou J, Ouzzine M (Março de 2007). «Substrate specificity of the human UDP-glucuronosyltransferase UGT2B4 and UGT2B7. Identification of a critical aromatic amino acid residue at position 33». The FEBS Journal. 274 (5): 1256–64. PMID 17263731. doi:10.1111/j.1742-4658.2007.05670.x
- ↑ Pathoma, Fundamentals of Pathology