Sequenciamento Shotgun
Em genética, o Sequenciamento Shotgun ou Sequenciação Shotgun, também conhecido como Clonagem Shotgun, é um método usado para sequenciamento de fitas longas de DNA. É batizado por analogia com o padrão de fogo de rápida expansão, quase aleatório de uma caçadeira (em inglês: shotgun).
O método de terminação de cadeia de sequenciamento de DNA (ou "Método de Sanger" por ter sido desenvolvido por Frederick Sanger) só pode ser usado para cadeias muito curtas de 100 a 1000 pares de bases. Sequências mais longas são subdivididas em fragmentos menores, que podem ser sequenciados separadamente, e, subsequentemente, elas são re-montadas para compor a sequência global. Dois métodos principais são usados para isso: primer walking (ou "chromosome walking") que progride através de toda a cadeia, pedaço por pedaço, e a Sequenciação Shotgun, que é um processo mais rápido, mas mais complexo que utiliza fragmentos aleatórios.
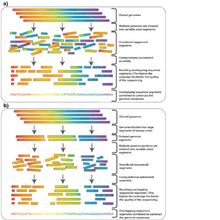
Na Sequenciação Shotgun,[1][2] o DNA é dividido aleatoriamente em numerosos pequenos segmentos, que são sequenciados utilizando o método de terminação de cadeia para obter leituras. Múltiplas leituras sobrepostas para o DNA-alvo são obtidas através da realização de várias rodadas desta fragmentação, e sequenciamento. Programas de computador, em seguida, usam as extremidades sobrepostas de diferentes leituras para reuni-las em uma sequência contínua.[1]
O sequenciamento Shotgun foi uma das tecnologias precursoras que foi responsável por possibilitar o sequenciamento do genoma completo.
Exemplo
editarPor exemplo, considere os seguintes duas rodadas de leituras shotgun:
| Cadeia | Sequência |
|---|---|
| Original | AGCATGCTGCAGTCATGCTTAGGCTA
|
| Primeira sequência shotgun | AGCATGCTGCAGTCATGCT--------------------------TAGGCTA
|
| Segunda sequência shotgun | AGCATG--------------------------CTGCAGTCATGCTTAGGCTA
|
| Reconstrução | AGCATGCTGCAGTCATGCTTAGGCTA
|
Neste exemplo extremamente simplificado, nenhuma das leituras cobrem o comprimento total da sequência original, mas as quatro leituras podem ser montadas na sequência original utilizando a sobreposição das suas extremidades para as alinhar e ordenar. Na realidade, esse processo usa quantidades enormes de informação que são repletas de ambiguidades e erros de sequenciamento. A montagem de genomas complexos é adicionalmente complicada pela grande abundância de sequências repetitivas, significando que leituras curtas semelhantes poderiam vir de partes completamente diferentes da sequência.
Referências
- ↑ a b Staden, R (1979). «A strategy of DNA sequencing employing computer programs». Nucleic Acids Research. 6 (7): 2601–10. PMC 327874 . PMID 461197. doi:10.1093/nar/6.7.2601
- ↑ Anderson, S (1981). «Shotgun DNA sequencing using cloned DNase I-generated fragments». Nucleic Acids Research. 9 (13): 3015–27. PMC 327328 . PMID 6269069. doi:10.1093/nar/9.13.3015
Ligações externas
editar- «Shotgun sequencing comes of age». The Scientist. Consultado em 31 de dezembro de 2002
- «Genomic shotgun sequencing». biology science. Consultado em 11 de abril de 2009. Arquivado do original em 12 de junho de 2013